先端医療研究センター血液・腫瘍研究部
Research Overview研究概要
加齢やがん化に伴う造血細胞の特質を理解して新規治療応用へ
高齢化に伴い二人に一人ががんになる時代です。2019年に発足した血液・腫瘍研究部は、ゲノム、エピゲノム、転写、スプライシング、代謝、細胞間クロストークの観点から未知のメカニズムを追求し、国内外の研究機関との連携ならびに産学官連携の上、治療応用へとつなげることを目標とします。特に急性骨髄性白血病(AML)や骨髄異形成症候群(MDS)など予後不良な悪性疾患に対して、腫瘍学の常識にとらわれず、様々な視点から「がん」を捉えることで新しいサイエンスの扉を開き医療へ還元することを目指します。
Memberメンバー
- 血液・腫瘍研究部長
- Professor
-
研究員
-
野村 真樹
Masaki Nomura
-
-
技術員
-
福本 未記
Miki Fukumoto
-
小池 優依
Yui Koike
-
伊藤 裕美
Hiromi Ito
-
-
大学院生(博士)
-
田中 淳
Atsushi Tanaka
-
肖 慕然
Muran Xiao
-
張 一帆
Yifan Zhang
-
臧 維嘉
Weijia Zang
-
青山 有美
Yumi Aoyama
-
雜賀 渉
Wataru Saika
-

- Research focus研究内容
- Publications発表論文
研究内容
がん細胞は正常細胞に比べて細胞増殖プログラム、そして、成熟した細胞に分化するプログラムの双方に異常が起きています。これらの異常の原因はがん細胞自体におこる遺伝子変異と考えられていますが、がん細胞が周囲の細胞からサポートを受けていることも明らかになっています。一つの遺伝子変異でがんが発症することは稀であり、複数の遺伝子配列の異常や遺伝子発現の制御機構の異常、代謝の変化、外部シグナルの変化などが複雑に絡み合ってがんを作り出しています。当研究室ではがんになる様々な原因について、ヒトデータに加えて動物モデルを用いて検証を重ね、がんをより深く理解し治療応用へとつなげることを目指します。とくに以下の点に注目をして研究をすすめています。
- (1) 転写後制御機構の異常に基づく発がん機構
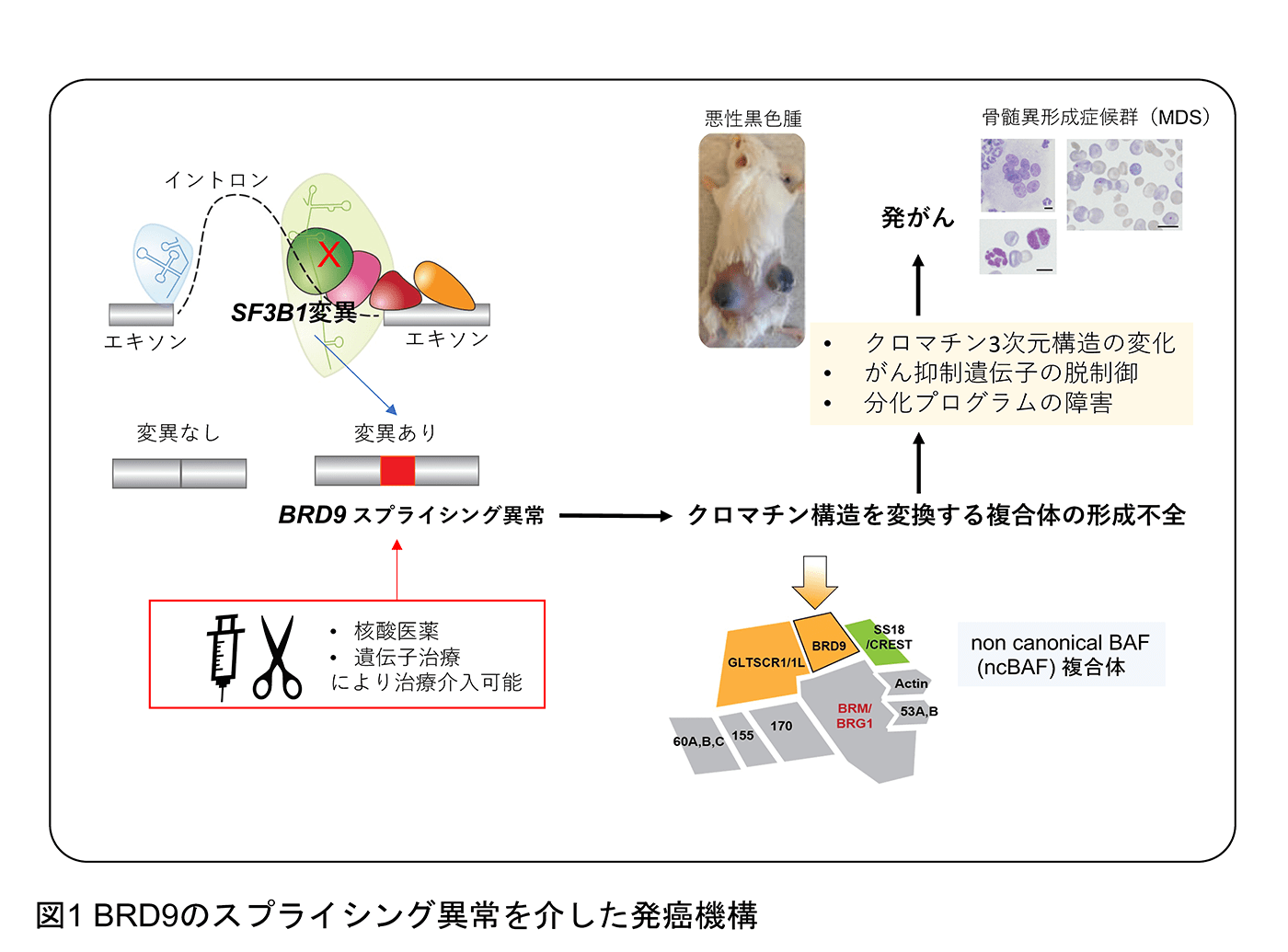
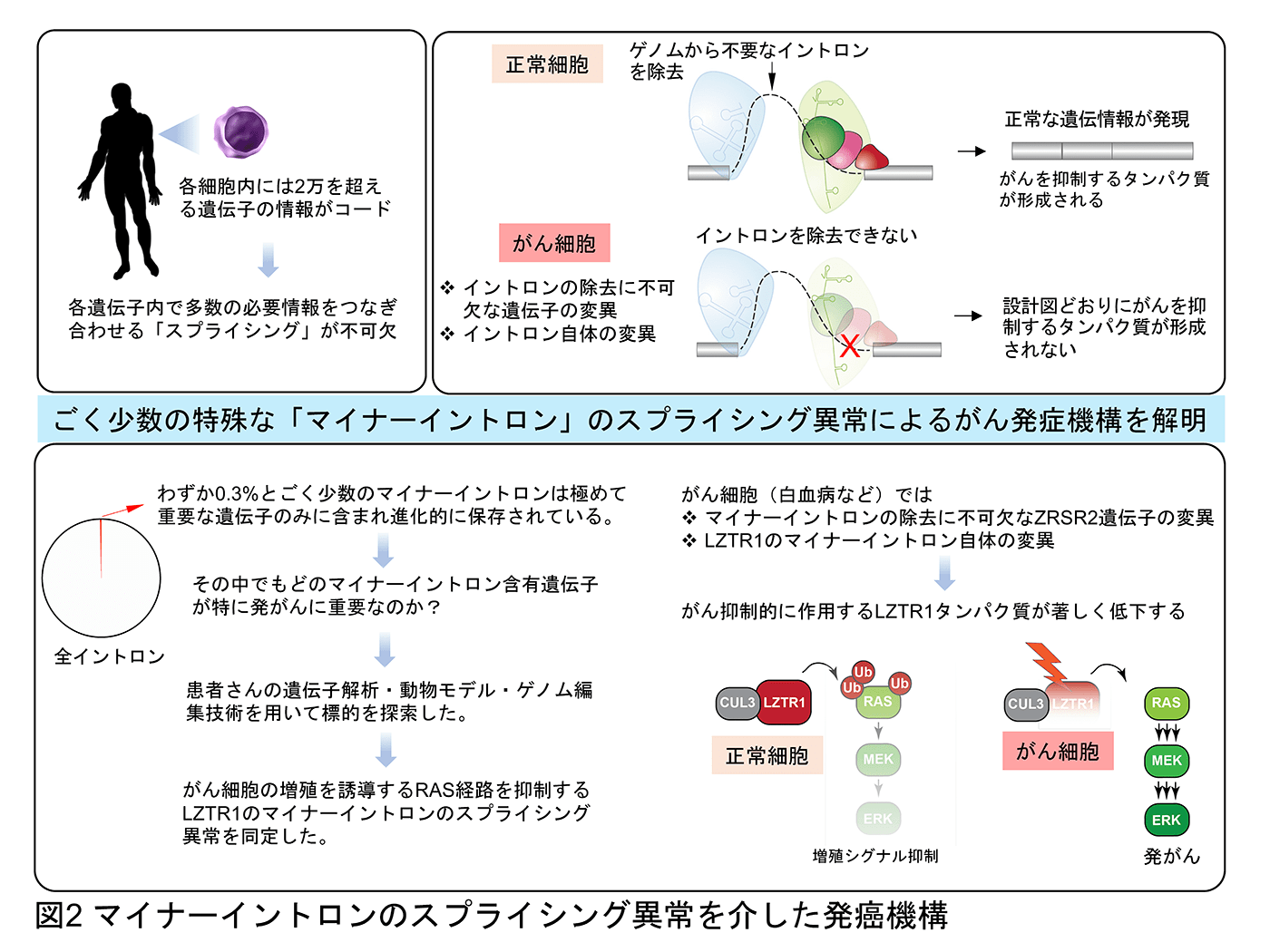
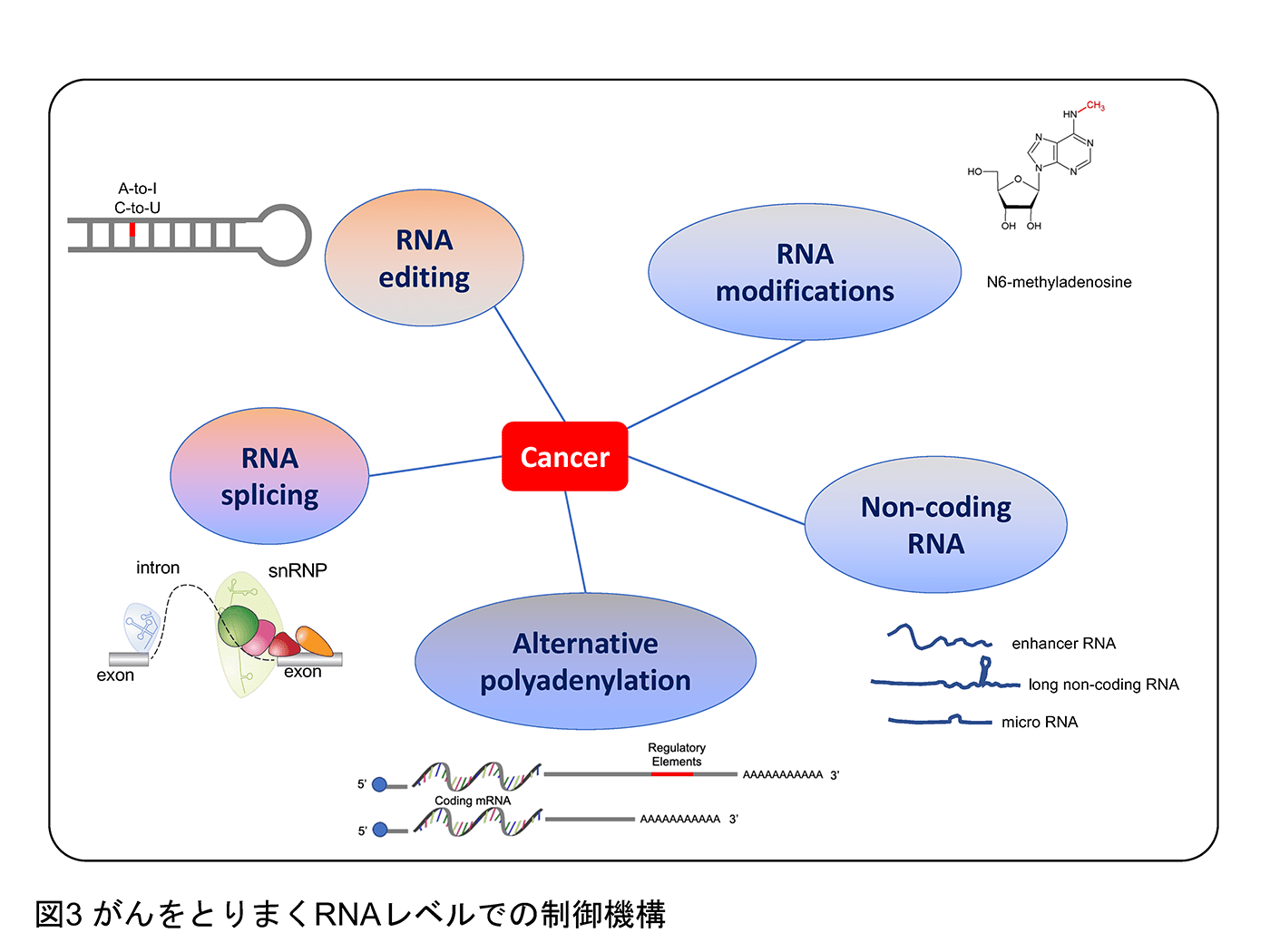
遺伝情報はDNAからpre-mRNAへと転写され、スプライシングを経て遺伝情報からイントロン などの不要な部分を取り除かれたmRNAがタンパクへと翻訳されます。このスプライシングをはじめとする「転写後制御」の過程で血液腫瘍を含む様々ながんで遺伝情報が歪められていくことをこれまでに報告してきました(Inoue et al. Nature 2019, Inoue et al. Nature Genetics 2021)。例えば、スプライシングに関連するSF3B1遺伝子の変異が、クロマチン(DNAとその周囲のタンパクの3次元構造)を制御するBRD9遺伝子の異常なスプライシングを引き起こし、BRD9タンパクの喪失を介して発がんを誘導します(Inoue et al. Nature 2019)。この点は、これまで別々に捉えられてきた遺伝子のスプライシングとクロマチンによる遺伝子発現制御をつなぐ新しい系路の発見につながりました(図1)。また、ごく少数でありながら進化的に重要な遺伝子のみに含有される「マイナーイントロン」の制御異常が発がんにつながることを報告してきました(Inoue et al. Nature Genetics 2021)(図2)。こちらもスプライシングの異常と発がんをつなげる興味深い知見であるだけでなく、進化の過程でなぜこのようなイントロンが保存されてきたのか、その性差がもたらす意義も含めて検討しています。また、スプライシングの異常メカニズムを解明することで、これまでに意義が不明であったイントロン変異に対してもスポットライトを当てることができます。イントロン変異によりスプライシングの異常を、ひいては発癌をきたす機構を明らかにしつつあり、治療応用を含めて研究を進めています。スプライシング以外にもRNAの段階で遺伝情報が書き換えられていることが近年次々と明らかとなっており(図3)、この制御機構の異常が発癌につながることが知られています。我々もRNAメチル化やRNAの核外輸送が正常細胞と腫瘍細胞でどのように違うのか、という点にも注目をして研究を進めています。
- (2) BRD9を標的とした白血病治療応用
上記のBRD9は急性骨髄性白血病においては、有望な治療標的となることが我々の研究で明らかとなっています。現在はBRD9タンパクを分解することにより白血病治療への応用、ならびに、協調的にはたらく薬剤の探索を同時におこなっています。BRD9はクロマチンの3次元構造の制御を介して造血幹細胞の運命制御に深く関与する遺伝子です。この点を利用して、メカニズムに応じた治療応用を試みています。
-
(3) 予後不良白血病のモデル作成とメカニズム解明
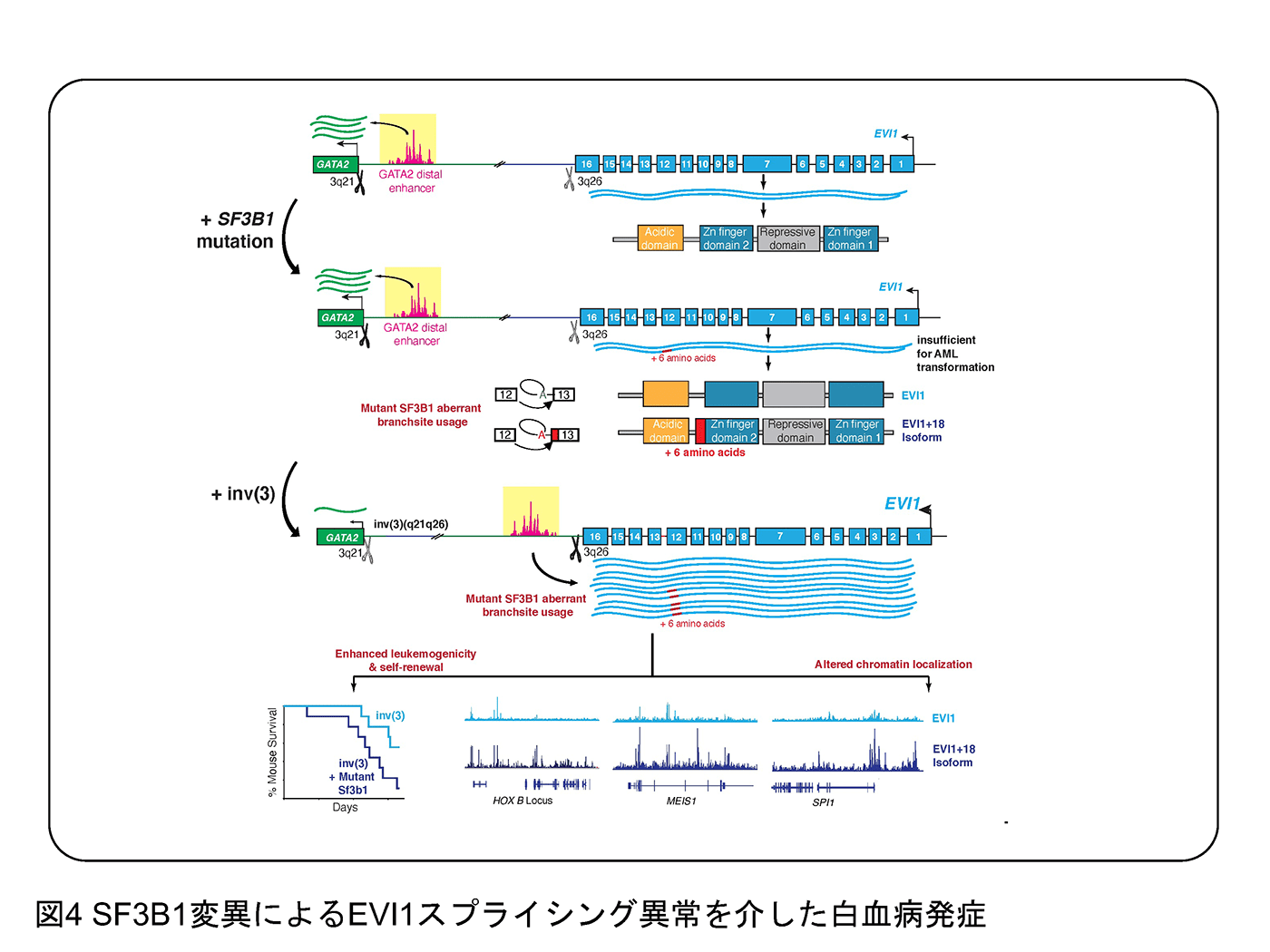
この20年間、急性骨髄性白血病(AML)の治療は、特定の亜集団においては大きな進歩を遂げてきましたが、まだまだ特異的な治療が見つからない急性骨髄性白血病では予後不良な疾患と言わざるをえません。我々はそれらのなかでも、3番染色体の逆位を有するAML、SETBP1変異を伴うAML、TP53変異を伴うAMLに特に注目をして機能解析を行っています。例えば、3番染色体の逆位を有するAMLにおいてはEVI1というAML幹細胞にとって重要な転写因子がエンハンサーハイジャッキングとよばれる現象によって高発現します。そのような症例では前述のSF3B1遺伝子の変異が高頻度に検出され、EVI1自体に異常なスプライシングが惹起され、新たなEVI1バリアントがより悪性度の高い白血病プログラムを誘導するという成果を報告しています(Tanaka et al. Blood 2022)(図4)。予後不良なAMLに対して、CRISPRスクリーニングなどのテクノロジーを用いて新しい治療戦略を提案できれば、と考えています。
-
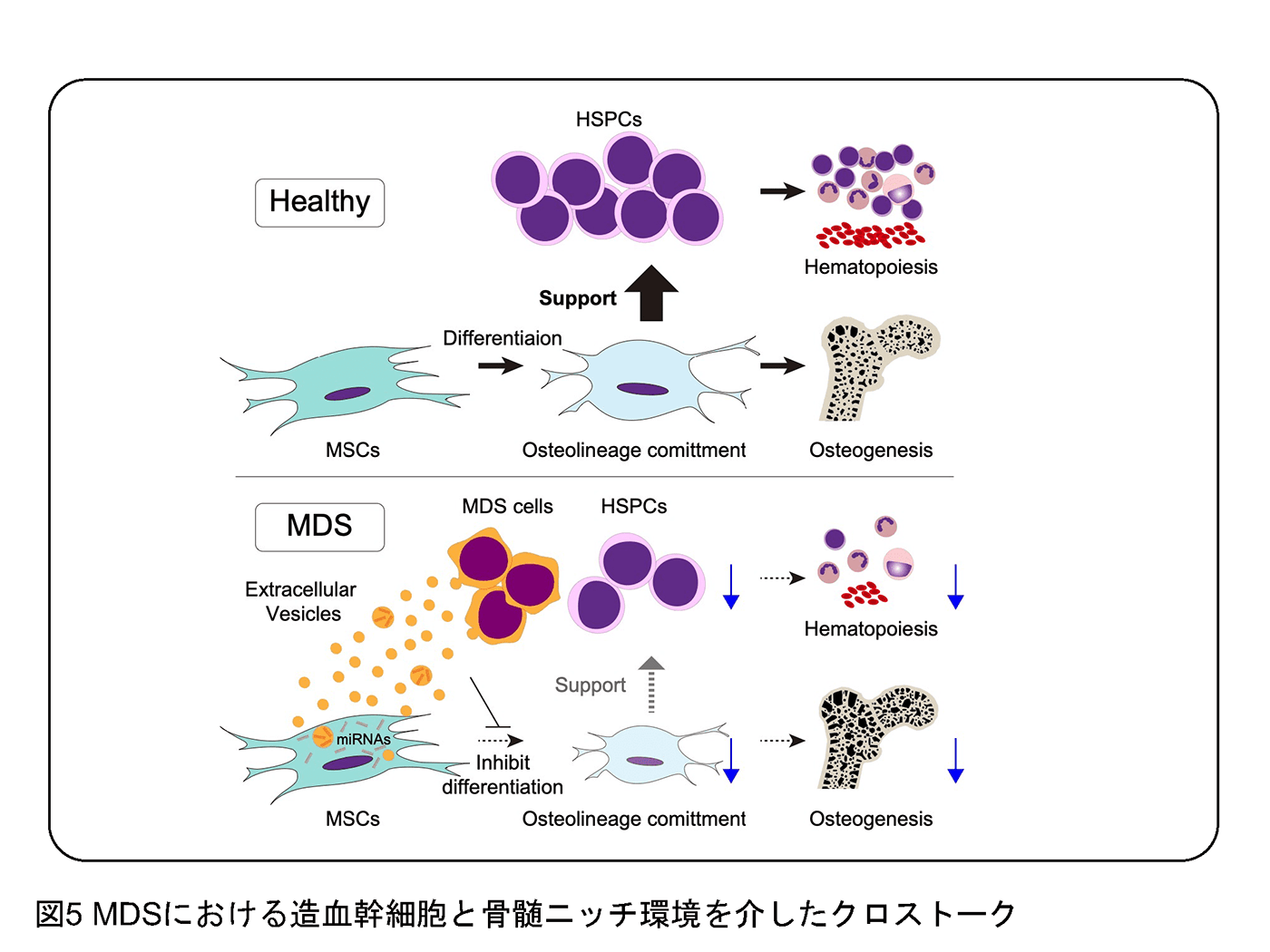
(4) 造血幹細胞と骨髄ニッチ環境との相互作用の解明
造血は骨髄という多細胞システムの中で生じます。すなわち、造血幹細胞自身の細胞自律的な制御だけでなく、周囲のニッチ環境、例えば、間葉系幹細胞、血管内皮細胞、神経細胞からの外的な制御を受けています。急性骨髄性白血病(AML)や骨髄異形成症候群(MDS)では、造血幹細胞に加えて周囲環境が大きく変化し、同時にその変化が腫瘍をさらに悪化させることがわかってきています。我々の研究では、MDSなどの異常な造血幹細胞が、周囲の造血環境を自身に有利にさせることで生存を担保しているという現象を捉えることができました。具体的には、腫瘍細胞由来のエキソソームが周囲の代表的なニッチ細胞である間葉系幹細胞に取り込まれることで、骨芽細胞系列への分化が抑制され、それによって正常造血への支持能が低下し、相対的にMDS細胞が有利になっていくという知見を報告しました(Hayashi et al. Cell Reports 2022)(図5)。このような研究をさらに発展させ、細胞間のクロストークや創薬につなげるべく研究をすすめています。
-
(5) 造血細胞に生じる新たな細胞死の意義
近年、細胞死には様々な種類があることが明らかになってきました。とりわけ、鉄イオンや脂質過酸化に伴う細胞死「フェロトーシス」と造血幹細胞の老化や発がん、そして治療応用に興味をもって研究をすすめています。障害を受けた造血幹細胞の抗酸化システムが正常と異なることを見出すことができれば、正しい病態理解と治療応用につながると期待されます。
この他にも、様々な視点から、造血細胞の「違い」に着目をして、研究を進めています。国内外の様々な研究室とコラボレーションしながらより奥行きのあるサイエンスを目指しています。我々と一緒に研究することを希望される方々がいらっしゃれば、是非お声がけいただければと思います。
業績
論文
- Chen S, Vedula RS, Cuevas-Navarro A, Lu B, Hogg SJ, Wang E, Benbarche S, Knorr K, Kim WJ, Stanley RF, Cho H, Erickson C, Singer M, Cui D, Tittley S, Durham BH, Pavletich TS, Fiala E, Walsh MF, Inoue D, Monette S, Taylor J, Rosen N, McCormick F, Lindsley RC, Castel P, Abdel-Wahab O. Impaired proteolysis of non-canonical RAS proteins drives clonal hematopoietic transformation. Cancer Discov. 2022 Oct 5;12(10):2434-2453.
- Nishimura K, Yamazaki H, Zang W, Inoue D. Dysregulated Minor Intron Splicing in Cancer. Cancer Sci. 2022 Sep;113(9):2934-2942.
- Tanaka A, Nakano AT, Nomura M, Yamazaki H, Bewersdorf JP, Lazaro RM, Hogg S, Liu B, Penson A, Yokoyama A, Zang W, Havermans M, Koizumi M, Hayashi Y, Cho H, Kanai A, Lee SC, Xiao M, Koike Y, Zhang Y, Fukumoto M, Aoyama Y, Konuma T, Kunimoto H, Inaba T, Nakajima H, Honda H, Kawamoto H, Delwel R, Abdel-Wahab O, Inoue D. Aberrant EVI1 splicing contributes to EVI1-rearranged leukemia. Blood, 2022 Jun 16:blood.2021015325. doi: 10.1182/blood.2021015325. Online ahead of print.
- Tanaka A, Nakano AT, Nomura M, Yamazaki H, Bewersdorf JP, Lazaro RM, Hogg S, Liu B, Penson A, Yokoyama A, Zang W, Havermans M, Koizumi M, Hayashi Y, Cho H, Kanai A, Lee SC, Xiao M, Koike Y, Zhang Y, Fukumoto M, Aoyama Y, Konuma T, Kunimoto H, Inaba T, Nakajima H, Honda H, Kawamoto H, Delwel R, Abdel-Wahab O, Inoue D. Aberrant EVI1 splicing contributes to EVI1-rearranged leukemia. Blood, 2022 Aug 25;140(8):875-888.
- Inoue D, Polaski JT, Taylor J, Castel P, Chen S, Kobayashi S, Hogg SJ, Hayashi Y, Bello Pineda JM, Ettaib EM, Erickson C, Knorr K, Fukumoto M, Yamazaki H, Tanaka A, Fukui C, Lu XL, Durham BH, Liu B, Wang E, Mehta S, Zakheim D, Grippa R, Penson A, Chew GL, McCormick F, Bradley RK, Abdel-Wahab O. Minor intron retention drives clonal hematopoietic disorders and diverse cancer predisposition. Nat Genetics, 2021 May;53(5):707-718.
- Rahmani NE, Ramachandra N, Sahu S, Gitego N, Lopez A, Pradhan K, Bhagat TD, Gordon-Mitchell S, Pena BR, Kazemi M, Rao K, Giricz O, Maqbool SB, Olea R, Zhao Y, Zhang J, Dolatshad H, Tittrea V, Tatwavedi D, Singh S, Lee J, Sun T, Steidl U, Shastri A, Inoue D, Abdel-Wahab O, Pellagatti A, Gavathiotis E, Boultwood J, Verma A. Blood Cancer J, 2021 Sep 21;11(9):157.
- Trivedi G, Inoue D, Zhang L. Targeting low-risk myelodysplastic syndrome with novel therapeutic strategies. Trends Mol Med. 2021 Oct;27(10):990-999.
- Fujino T, Goyama S, Sugiura Y, Inoue D, Asada S, Yamasaki S, Matsumoto A, Yamaguchi K, Isobe Y, Tsuchiya A, Shikata S, Sato N, Morinaga H, Fukuyama T, Tanaka Y, Fukushima T, Takeda R, Yamamoto K, Honda H, Nishimura E, Furukawa Y, Shibata T, Abdel-Wahab O, Suematsu M, Kitamura T. Mutant ASXL1 induces age-related expansion of phenotypic hematopoietic stem cells through activation of Akt/mTOR pathway. Nat Commun, Mar 23;12(1):1826. doi: 10.1038/s41467-021-22053-y..
- HHEX promotes myeloid transformation in cooperation with mutant ASXL1. Takeda R, Asada S, Park SJ, Yokoyama A, Becker HJ, Kanai A, Visconte V, Hershberger CE, Hayashi Y, Yonezawa T, Tamura M, Fukushima T, Tanaka Y, Fukuyama T, Matsumoto A, Yamasaki S, Nakai K, Yamazaki S, Inaba T, Shibata T, Inoue D, Honda H, Goyama S, Maciejewski JP, Kitamura T. Blood, 2020 Oct 1;136(14):1670-1684.
- Inoue D, Chew GL, Liu B, Michel BC, Pangallo J, D’Avino AR, Hitchman T, North K, Lee SC, Bitner L, Ariele B, Moore AR, Yoshimi A, Hoyos LE, Cho H, Penson A, Lu SX, Taylor J, Chen Y, Kadoch C, Abdel-Wahab O, Bradley RK. Spliceosomal disruption of the non-canonical BAF complex in cancer. Nature, 2019 Oct;574(7778):432-436.
- Yoshimi A, Lin KT, Wiseman DH, Rahman MA, Pastore1 A, Intlekofer AM, Wang B, Lee SCW, Micol JB, Zhang XJ, Inoue D, Thompson CB, Levine RL, Bradley RK, Abdel-Wahab O. Coordinated Alterations in RNA Splicing and Epigenetic Regulation Drive Leukemogenesis. Nature, 2019 Oct;574(7777):273-277.
- Trivedi G, Inoue D, Chen C, Bitner L, Chung YR, Justin T, Gönen M, Wess J, Abdel-Wahab O, and Zhang L. Muscarinic acetylcholine receptor regulates self-renewal of early erythroid progenitors. Sci Transl Med. 2019 Sep 25;11(511).
- 12. Taylor J, Sendino M, Gorelick AN, Pastore A, Chang MT, Penson AV, Gavrila EI, Stewart C, Melnik EM, Herrejon Chavez F, Bitner L, Yoshimi A, Lee SC, Inoue D, Liu B, Zhang XJ, Mato AR, Dogan A, Kharas MG, Chen Y, Wang D, Soni RK, Hendrickson RC, Prieto G, Rodriguez JA, Taylor BS, Abdel-Wahab O. Altered nuclear export signal recognition as a driver of oncogenesis. Cancer Discov, 2019 Oct;9(10):1452-1467.
- Wang E, Lu SX, Pastore A, Chen X, Imig J, Chun-Wei Lee S, Hockemeyer K, Ghebrechristos YE, Yoshimi A, Inoue D, Ki M, Cho H, Bitner L, Kloetgen A, Lin KT, Uehara T, Owa T, Tibes R, Krainer AR, Abdel-Wahab O, Aifantis I. Targeting an RNA-Binding Protein Network in Acute Myeloid Leukemia. Cancer Cell. 2019 Mar 18;35(3):369-384.
- Saika M, Inoue D, Nagase R, Sato N, Tsuchiya A, Yabushita T, Kitamura T, Goyama S. ASXL1 and SETBP1 mutations promote leukaemogenesis by repressing TGFβ pathway genes through histone deacetylation. Sci Rep. 2018 Oct 26;8(1):15873.
- Lee SC, North K, Kim E, Jang E, Obeng E, Lu SX, Liu B, Inoue D, Yoshimi A, Ki M, Yeo M, Zhang XJ, Kim MK, Cho H, Chung YR, Taylor J, Durham BH, Kim YJ, Pastore A, Monette S, Palacino J, Seiler M, Buonamici S, Smith PG, Ebert BL, Bradley RK, Abdel-Wahab O. Synthetic Lethal and Convergent Biological Effects of Cancer-Associated Spliceosomal Gene Mutations. Cancer Cell. 2018 Aug 34(2):225-241.
- Asada S, Goyama S, InoueD, Shikata S, Takeda R, Fukushima T, Yonezawa T, Fujino T, Hayashi Y, Kawabata KC, Fukuyama T, Tanaka Y, Yokoyama A, Yamazaki S, Kozuka-Hata H, Oyama M, Kojima S, Kawazu M, Mano H, Kitamura T. Mutant ASXL1 cooperates with BAP1 to promote myeloid leukaemogenesis. Nat Commun. 2018 Jul16;9(1):2733.
- Taylor J, Pavlick D, Yoshimi A, Marcelus C, Chung SS, Hechtman JF, Benayed R, Cocco E, Durham BH, Bitner L, Inoue D, Chung YR, Mullaney K, Watts JM, Diamond EL, Albacker LA, Mughal TI, Ebata K, Tuch BB, Ku N, Scaltriti M, Roshal M, Arcila M, Ali S, Hyman DM, Park JH, Abdel-Wahab O. Oncogenic TRK fusions are amenable to inhibition in hematologic malignancies. J Clin Invest. 2018 Aug 31;128(9):3819-3825.
- Nagase R, Inoue D (co-first and Corresponding author), Pastore A, Fujino T, Hou HA, Yamasaki N, Goyama S, Saika M, Kanai A, Sera Y, Horikawa S, Ota Y, Asada S, Hayashi Y, Kawabata KC, Takeda R, Tien HF, Honda H, Abdel-Wahab O, Kitamura T. Expression of mutant Asxl1 perturbs hematopoiesis and promotes susceptibility to leukemic transformation. J Exp Med. 2018 Jun 215(6):1729-1747.
- Inoue D (Corresponding author), Fujino T, Sheridan P, Zhang YZ, Nagase R, Horikawa S, Li Z, Matsui H, Kanai A, Saika M, Yamaguchi R, Kozuka-Hata H, Kawabata KC, Yokoyama A, Goyama S, Inaba T, Imoto S, Miyano S, Xu M, Yang FC, Oyama M, Kitamura T. A novel ASXL1-OGT axis plays roles in H3K4 methylation and tumor suppression in myeloid malignancies. Leukemia. 2018 Jun 32(6):1327-1337.
- Kawabata KC, Hayashi Y, Inoue D, Meguro H, Sakurai H, Fukuyama T, Tanaka Y, Asada S, Fukushima T, Nagase R, Takeda R, Harada Y, Kitaura J, Goyama S, Harada H, Aburatani H, Kitamura T. High expression of ABCG2 induced by EZH2 disruption plays pivotal roles in MDS pathogenesis. Leukemia. 2018 Feb 32(2):419-428.
- Inoue D, Fujino T, Kitamura T. ASXL1 as a critical regulator of epigenetic marks and therapeutic potential of mutated cells. LOncotarget. 2018 Oct 16;9(81):35203-35204.
- Micol JB, Pastore A, Inoue D (co-first author), Duployez N, Kim E, Lee SC, Durham BH, Chung YR, Cho H, Zhang XJ, Yoshimi A, Krivtsov A, Koche R, Solary E, Sinha A, Preudhomme C, Abdel-Wahab O. ASXL2 is essential for haematopoiesis and acts as a haploinsufficient tumour suppressor in leukemia. Nat Commun. 2017 May 18;8:15429, 2017.
- Inoue D, Abdel-Wahab O. Modeling SF3B1 Mutations in Cancer: Advances, Challenges, and Opportunities. Cancer Cell. 2016 Sep 12;30(3):371-373.
- Inoue D, Bradley RK, Abdel-Wahab O. Spliceosomal gene mutations in myelodysplasia: molecular links to clonal abnormalities of hematopoiesis. Genes Dev. 2016 May 1;30(9):989-1001.
日本語総説
- 張一帆、肖慕然、臧維嘉、井上大地. 造血器腫瘍におけるマイナーイントロンリテンションの意義、科学評論社、血液内科、pp681-686、2021
- 村上賢、山嵜博未、林康貴、井上大地. クローン性造血とMDS. メディカルサイエンスダイジェスト、ニューサイエンス社、東京、pp22-25、2021.
- 小林漸、肖慕然、田中淳、井上大地. 骨髄系腫瘍におけるスプライシング異常と治療応用.
血液内科、科学評論社、東京、pp581-586、2020. - 井上大地. がん幹細胞と転写後制御. 医学のあゆみ、医歯薬出版、東京、pp375-379、2020.
- 井上大地. スプライシング異常を原因とした新しい発がん機構の解明と治療応用. 実験医学、羊土社、東京、38:451-455, 2020.
- 田中淳、小林漸、肖慕然、井上大地.スプライシング異常に基づく発がん機構の解明と治療応用、臨床血液, 61:643-650, 2020.v
- 井上大地. スプライソソーム異常が拓く造血器腫瘍研究の未来. 実験医学、羊土社、東京、pp3084-3089、2017.
近年の主な受賞歴
(井上 大地)
- 麒麟児賞、麒麟塾
- 2021年度MSD生命科学財団医学奨励賞最優秀賞(がん領域)、MSD生命科学財団
- 井上リサーチアウォード、井上科学振興財団
- アステラス病態代謝研究会優秀発表賞、アステラス病態代謝研究会
- UJA Outstanding Paper Award、United Japanese Researchers Around the World
- 神戸医療産業都市推進機構先端医療研究センター長賞、神戸医療産業都市推進機構
- 神戸医療産業都市推進機構理事長賞、神戸医療産業都市推進機構/
- ASH (American Society of Hematology) Global Research Award、米国血液学会
- 荻村孝特別研究賞、日本白血病研究基金
- Young Investigator Award, MDS Foundation、米国





